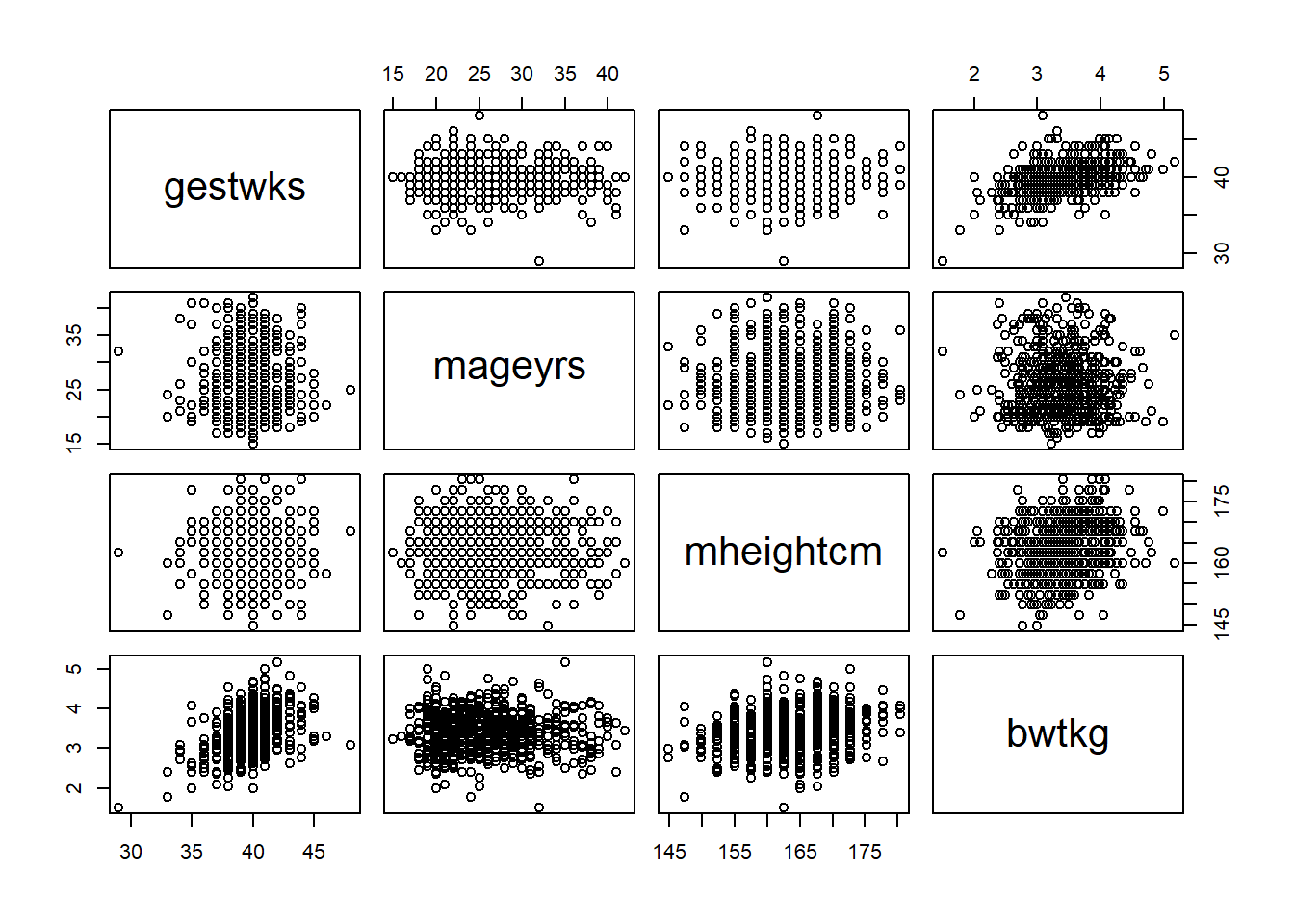
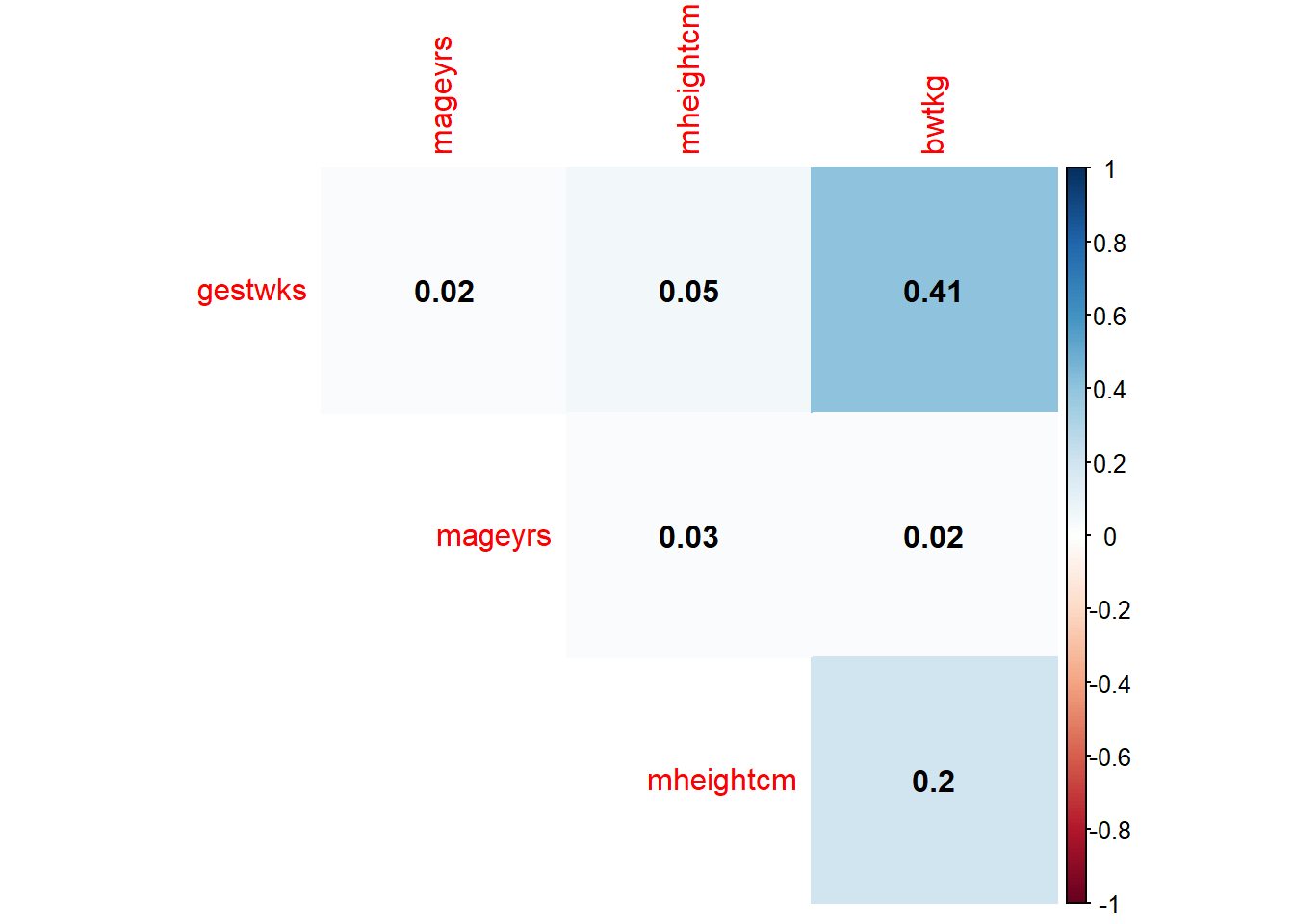
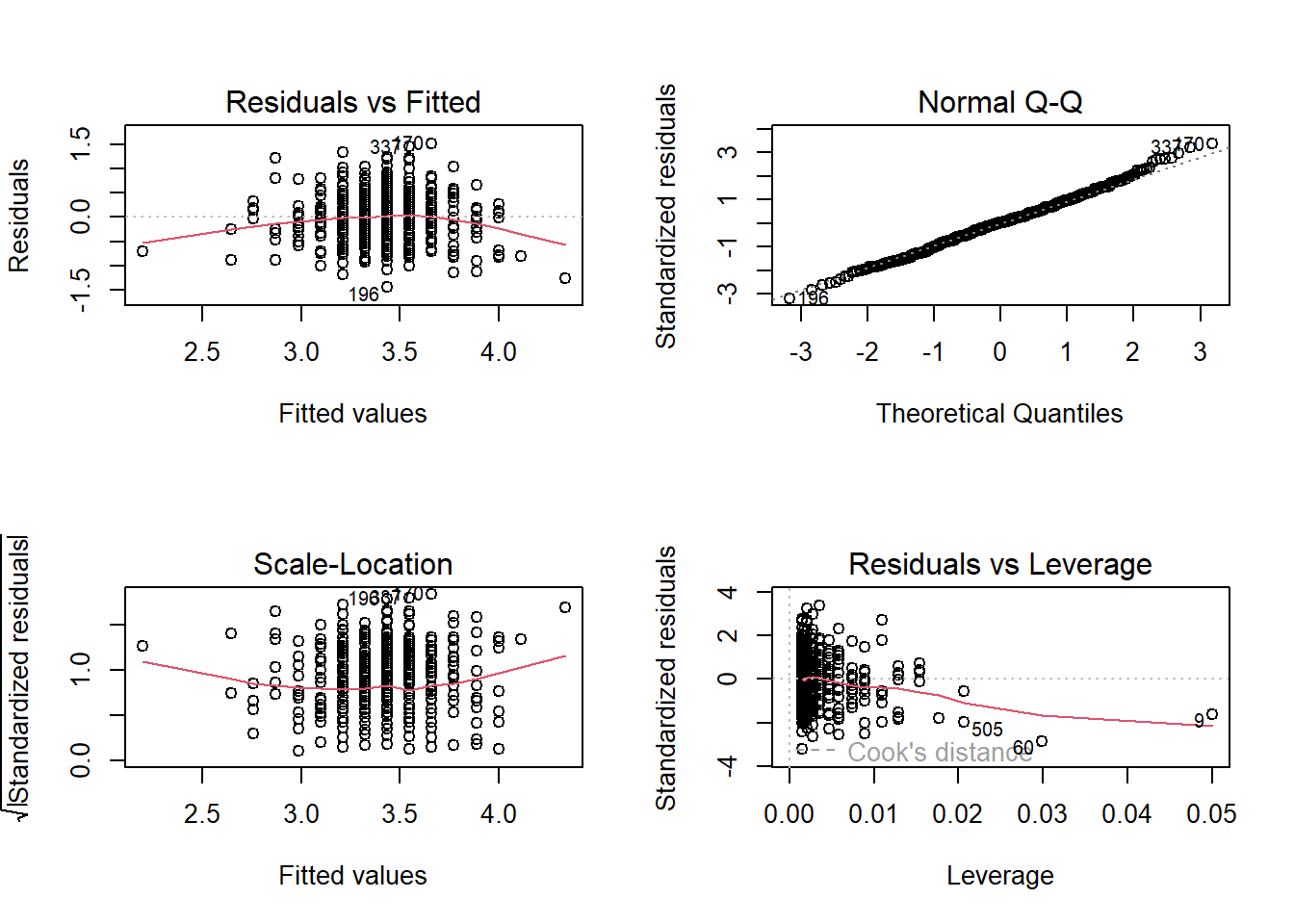
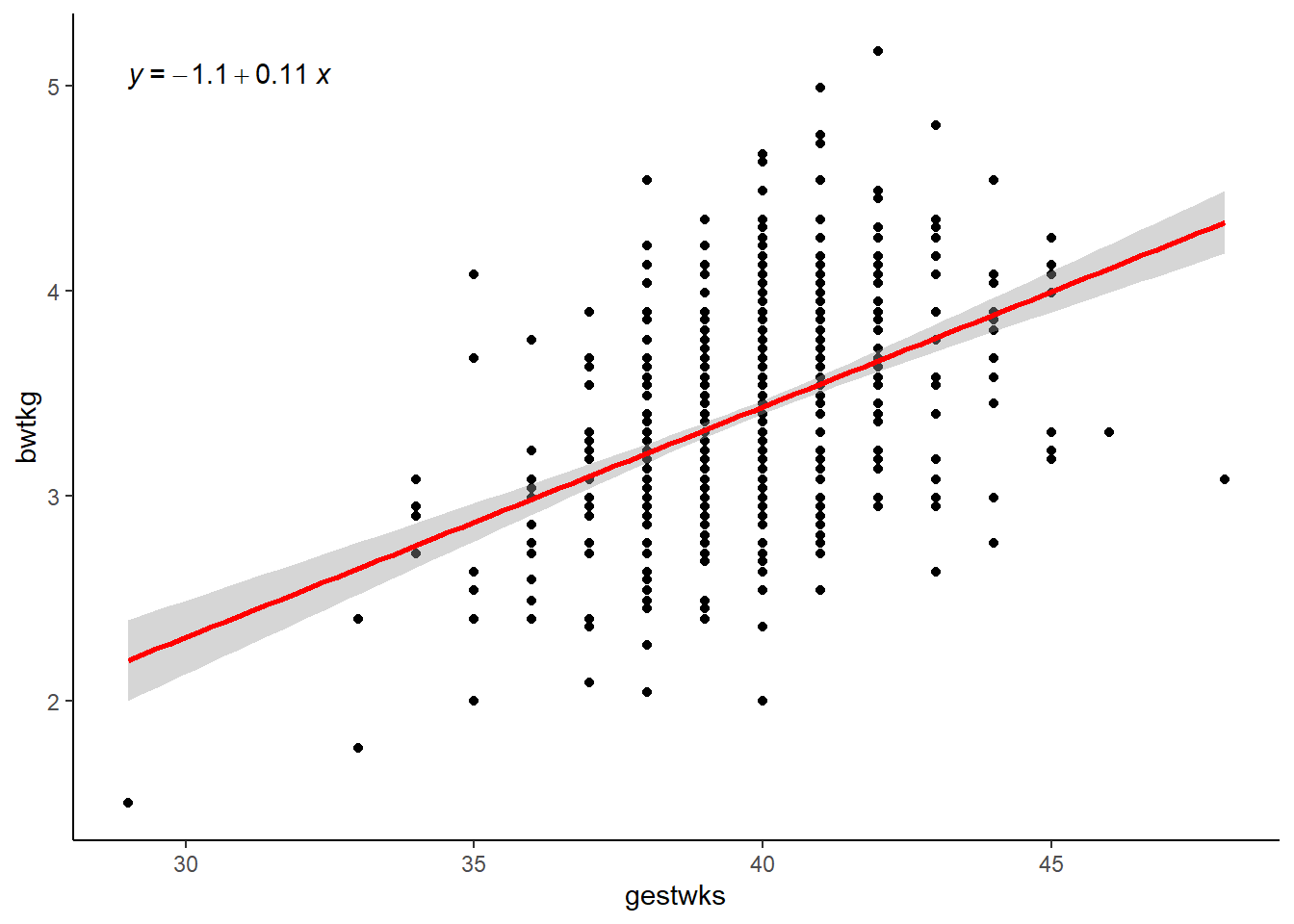
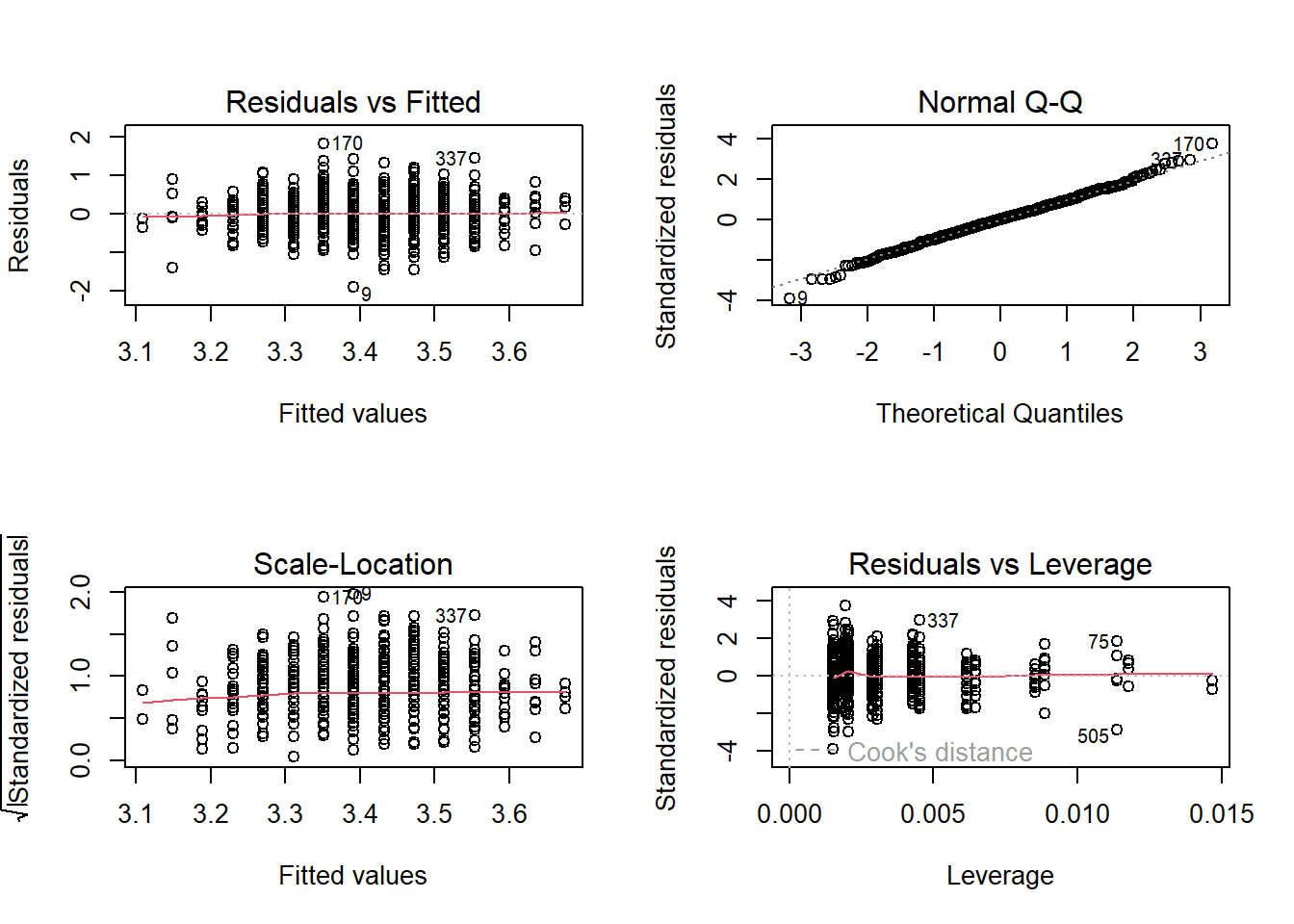
Rows: 680
Columns: 13
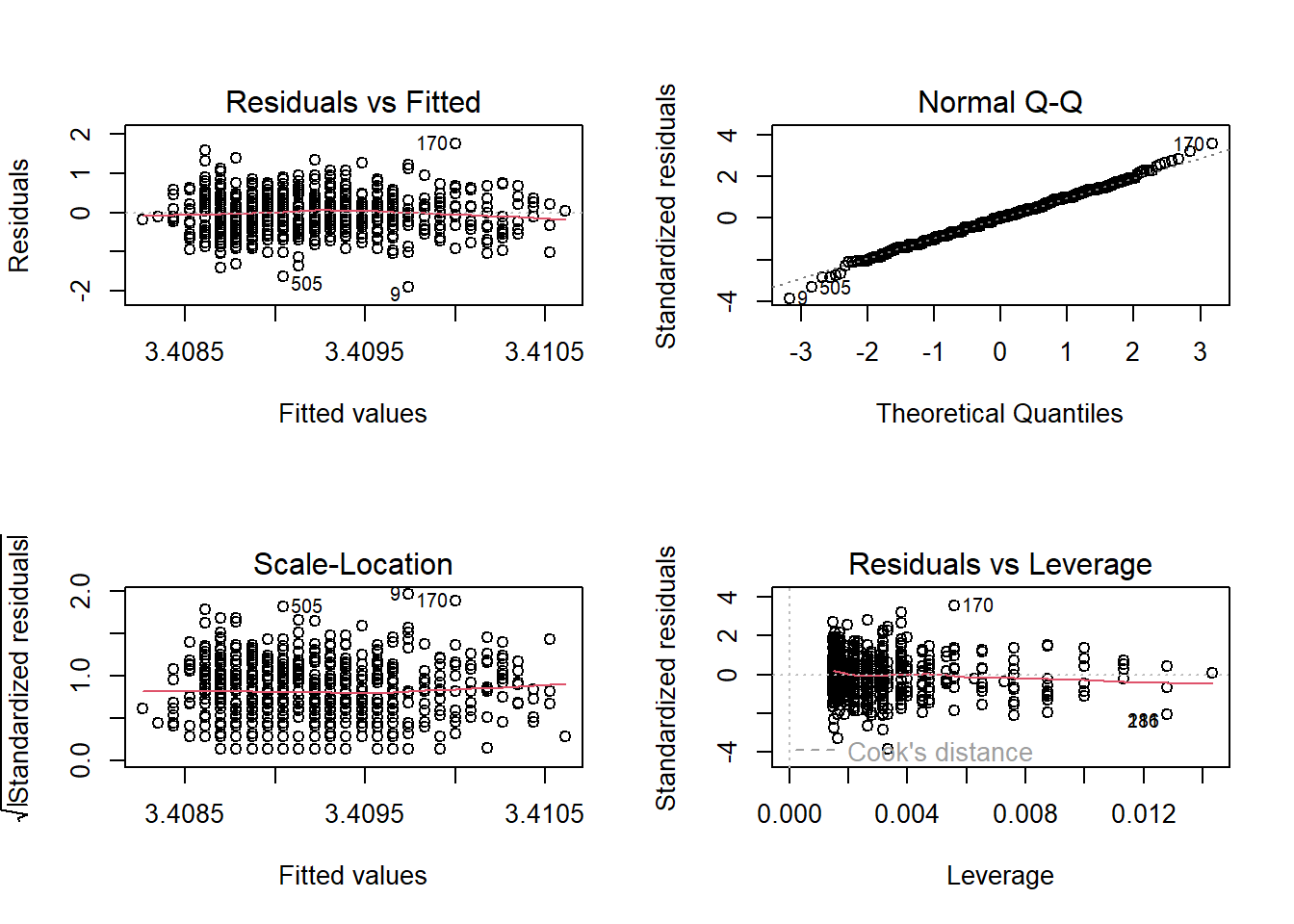
$ bwtkg <dbl> 3.31, 3.63, 3.40, 3.18, 2.40, 3.90, 4.13, 2.95, 1.50, 3.67,…
$ blengthcm <dbl> 50.80, 53.34, 53.34, 50.80, 48.26, 50.80, 55.88, 48.26, 50.…
$ bheadcircm <dbl> 33.02, 33.02, 33.02, 33.02, 33.02, 35.56, 38.10, 33.02, 30.…
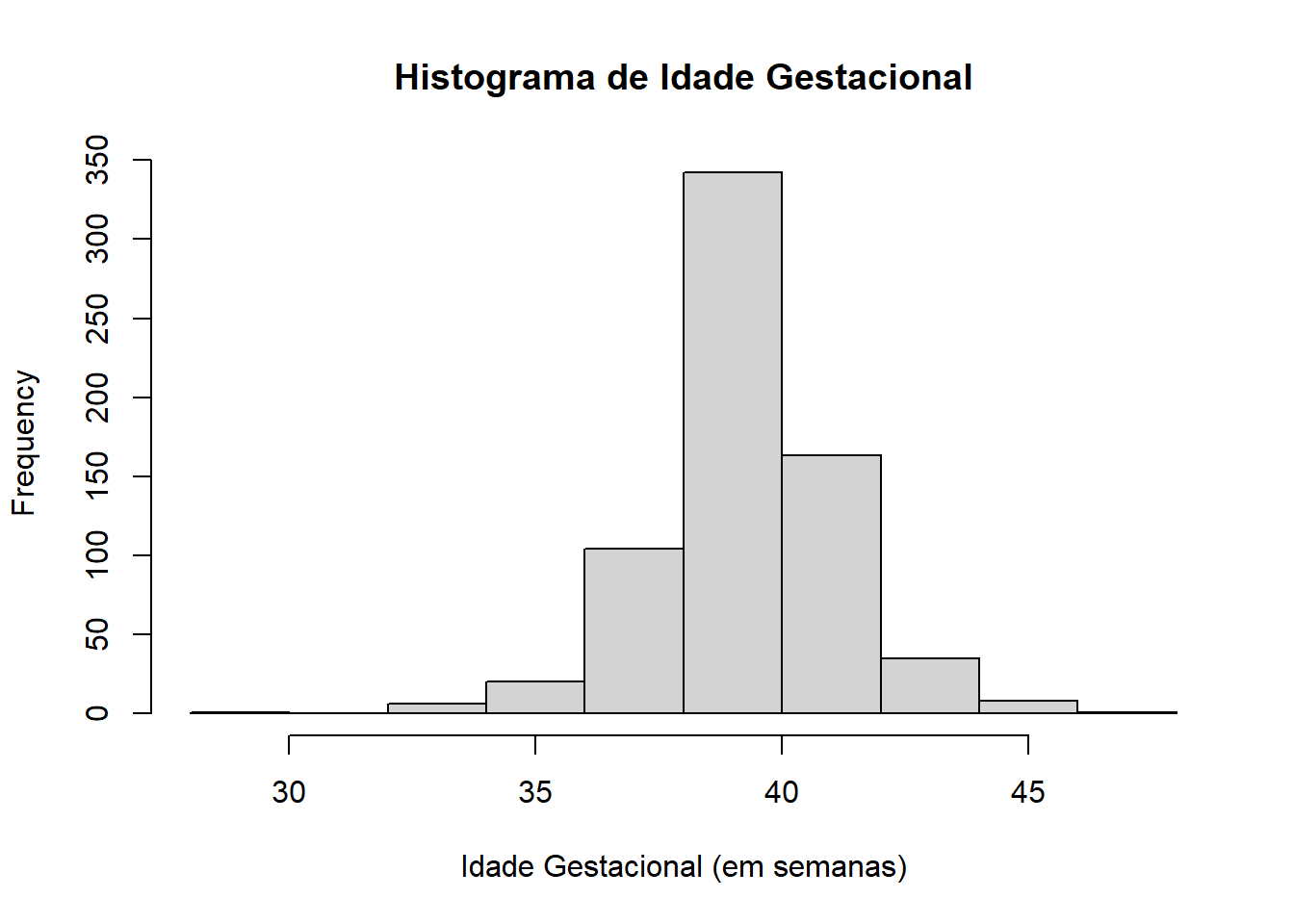
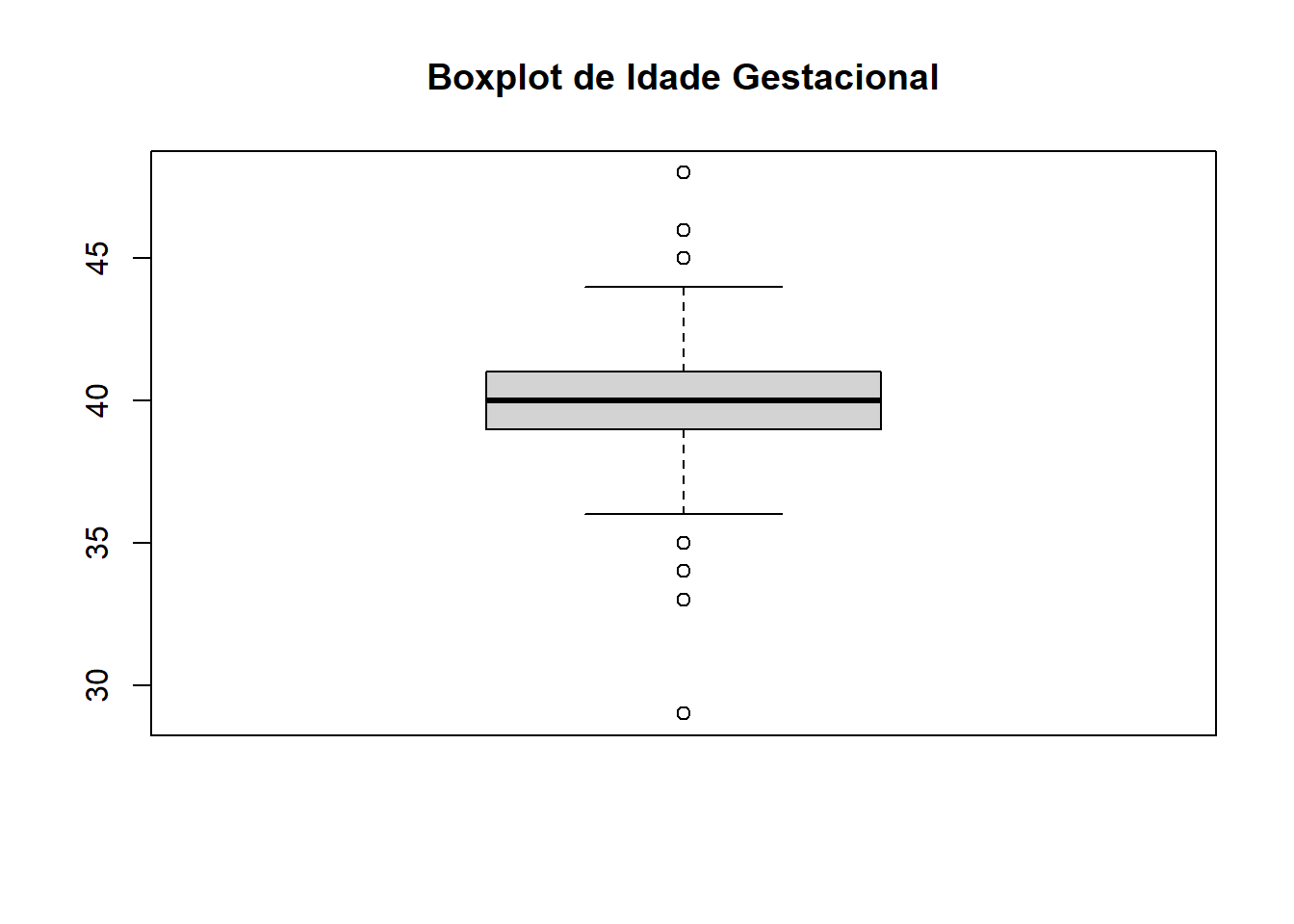
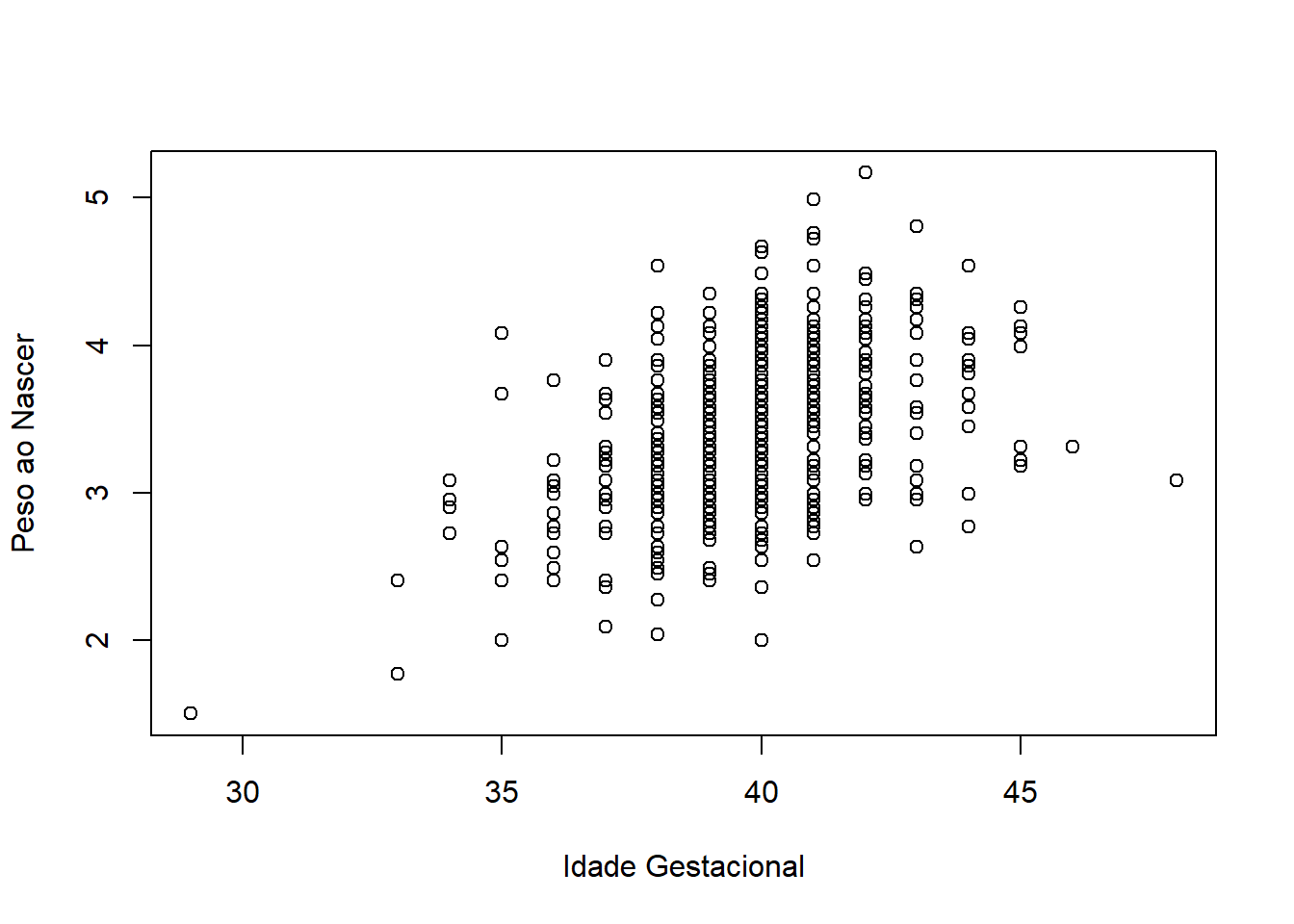
$ gestwks <dbl> 37, 41, 39, 39, 37, 43, 40, 37, 29, 41, 40, 39, 41, 41, 42,…
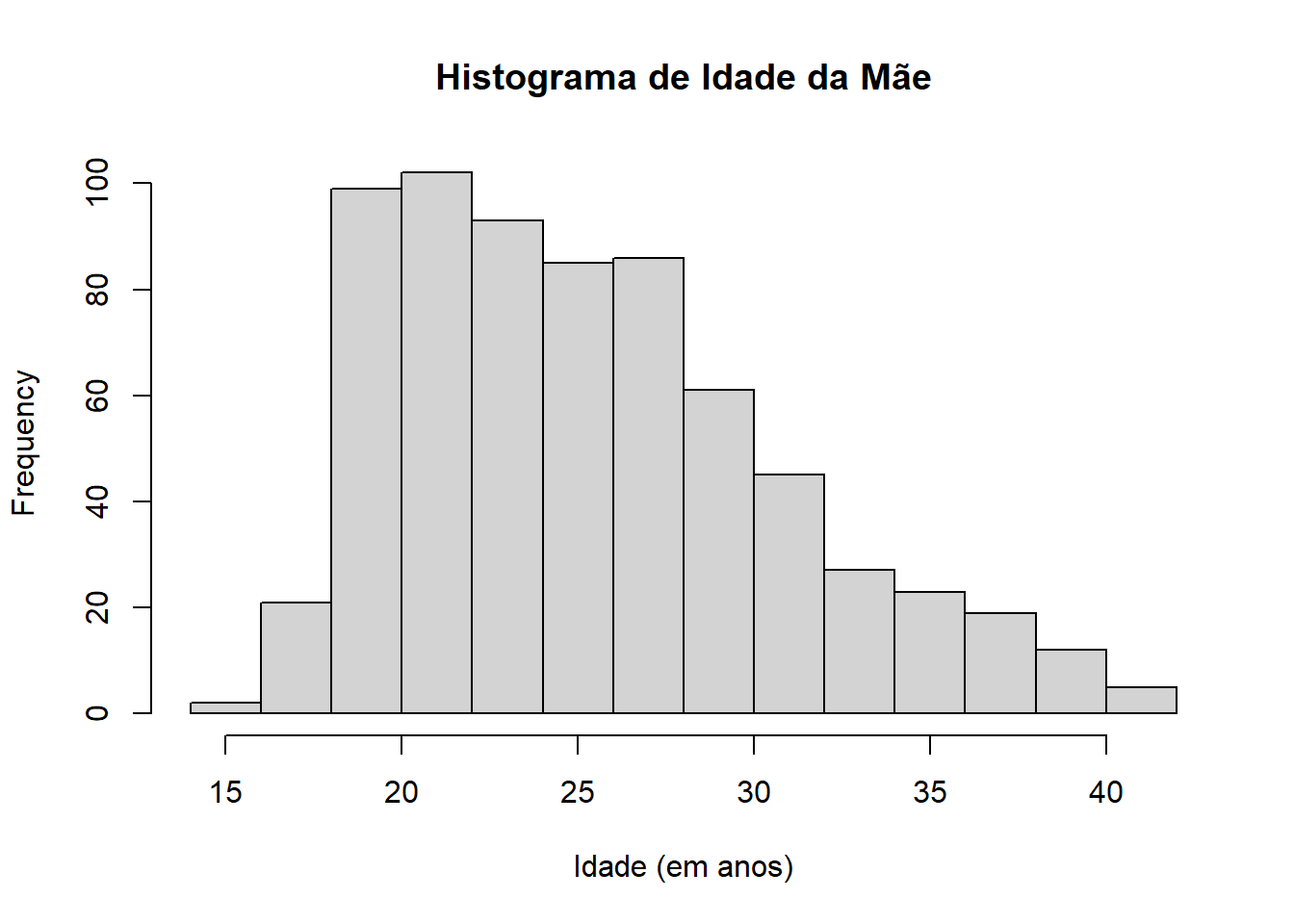
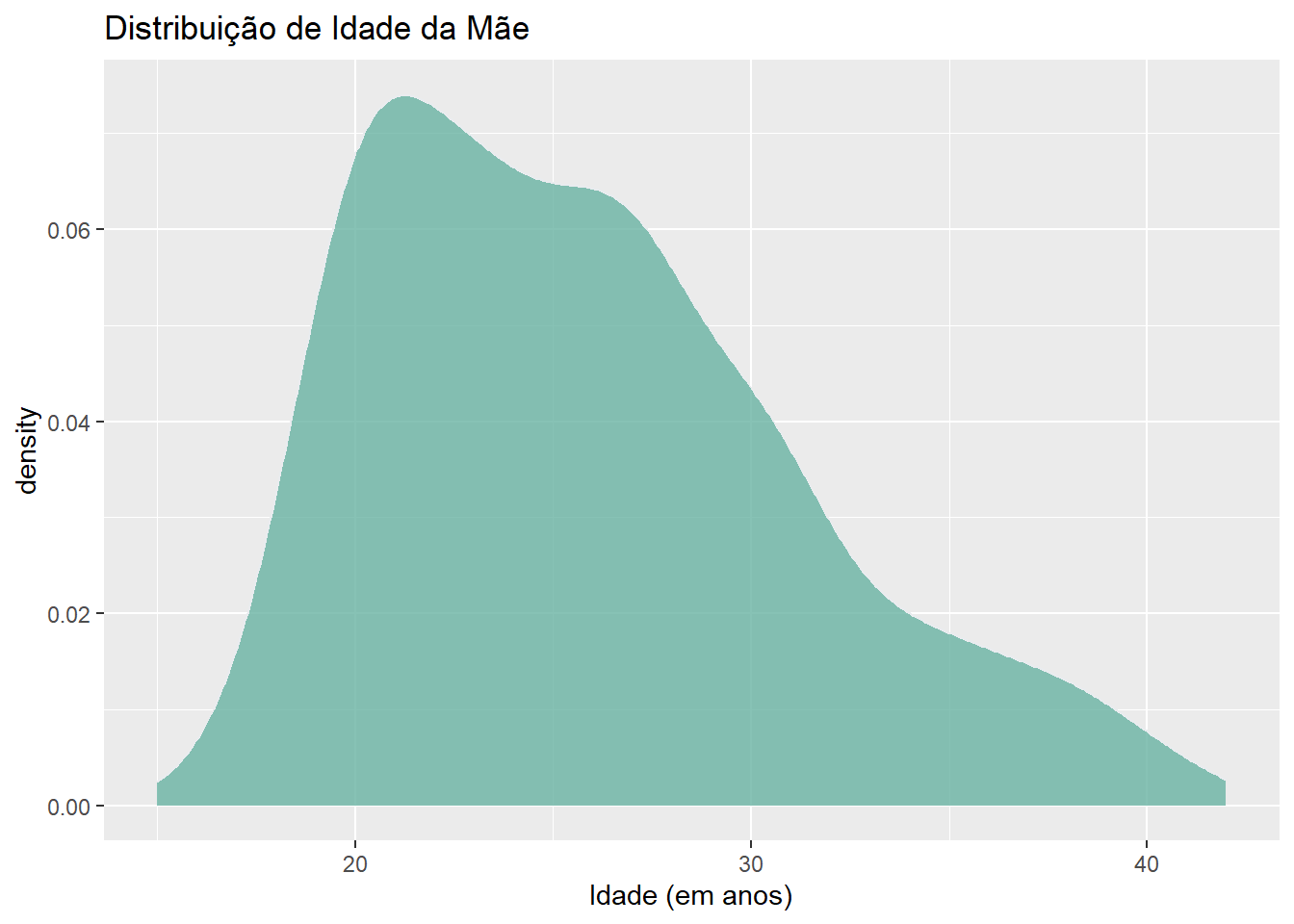
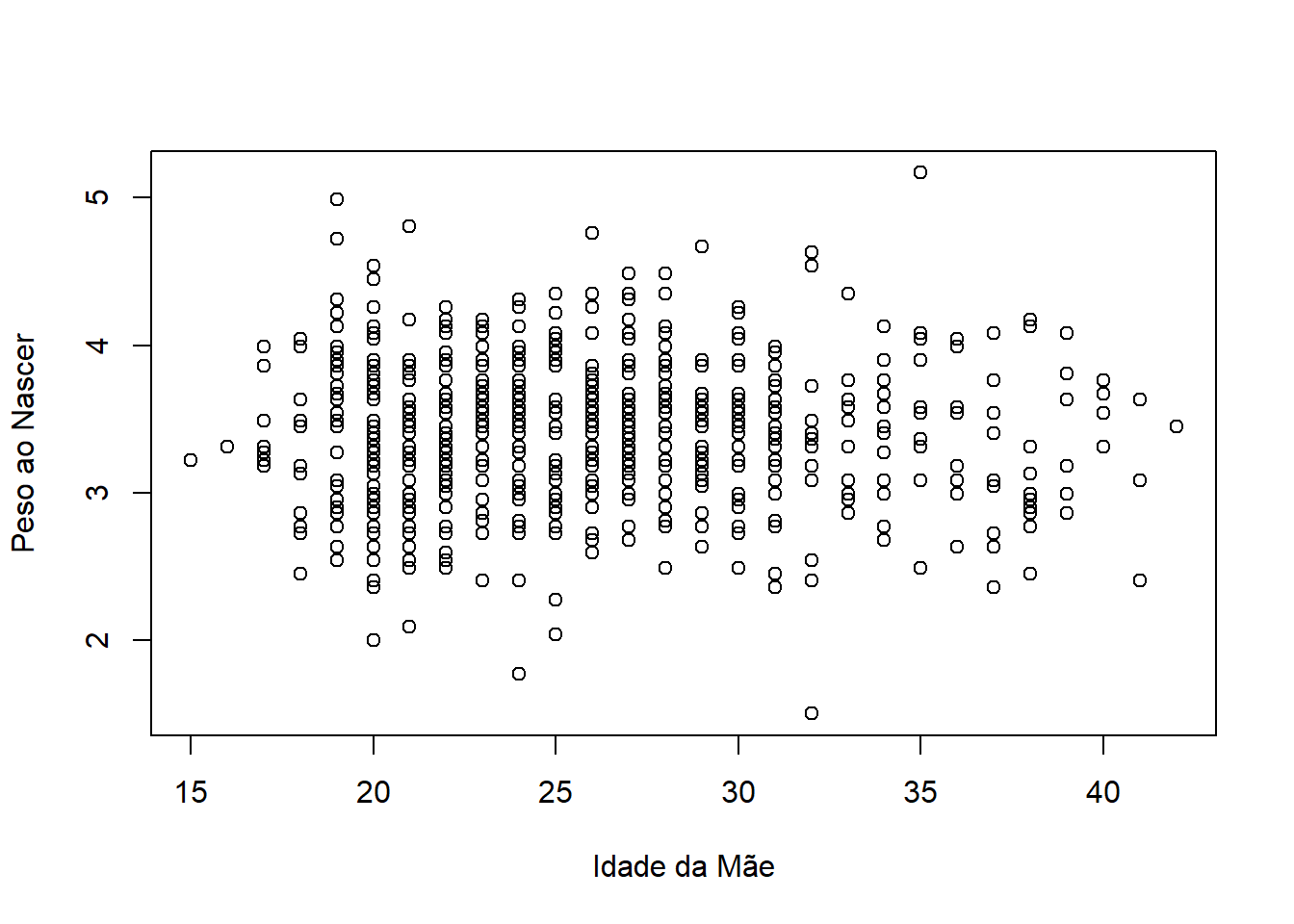
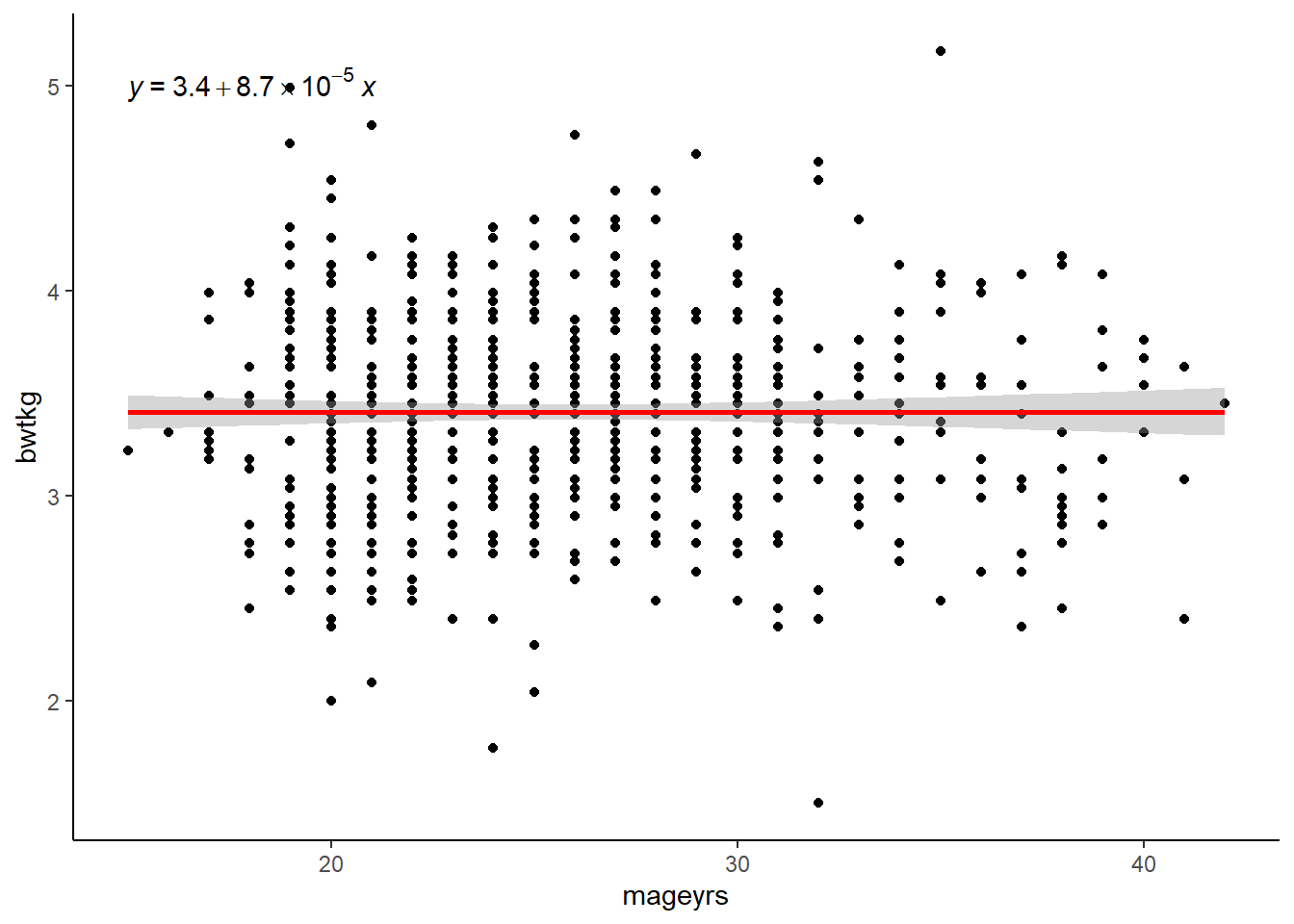
$ mageyrs <dbl> 33, 28, 32, 27, 32, 30, 23, 27, 32, 28, 26, 19, 37, 31, 29,…
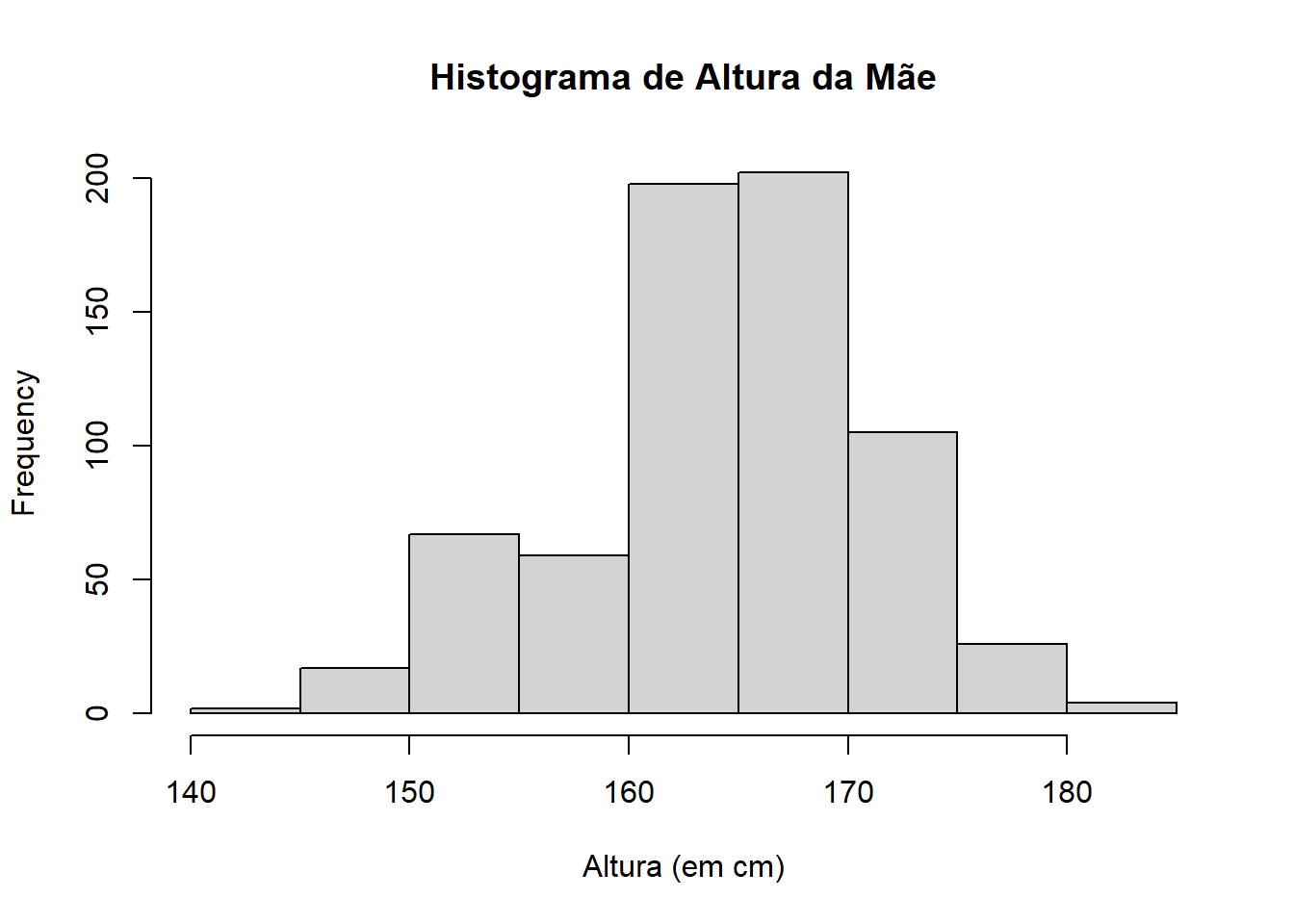
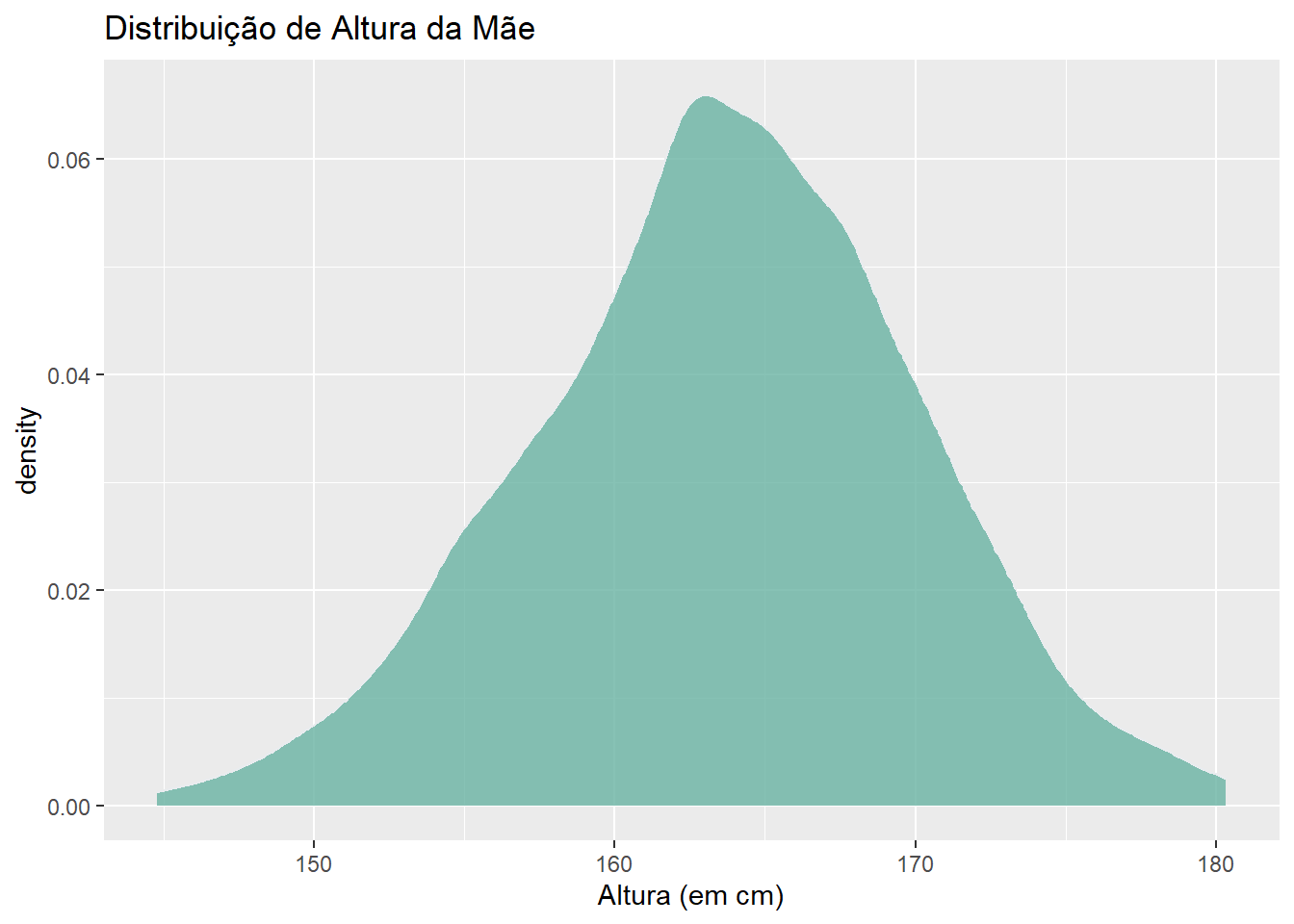
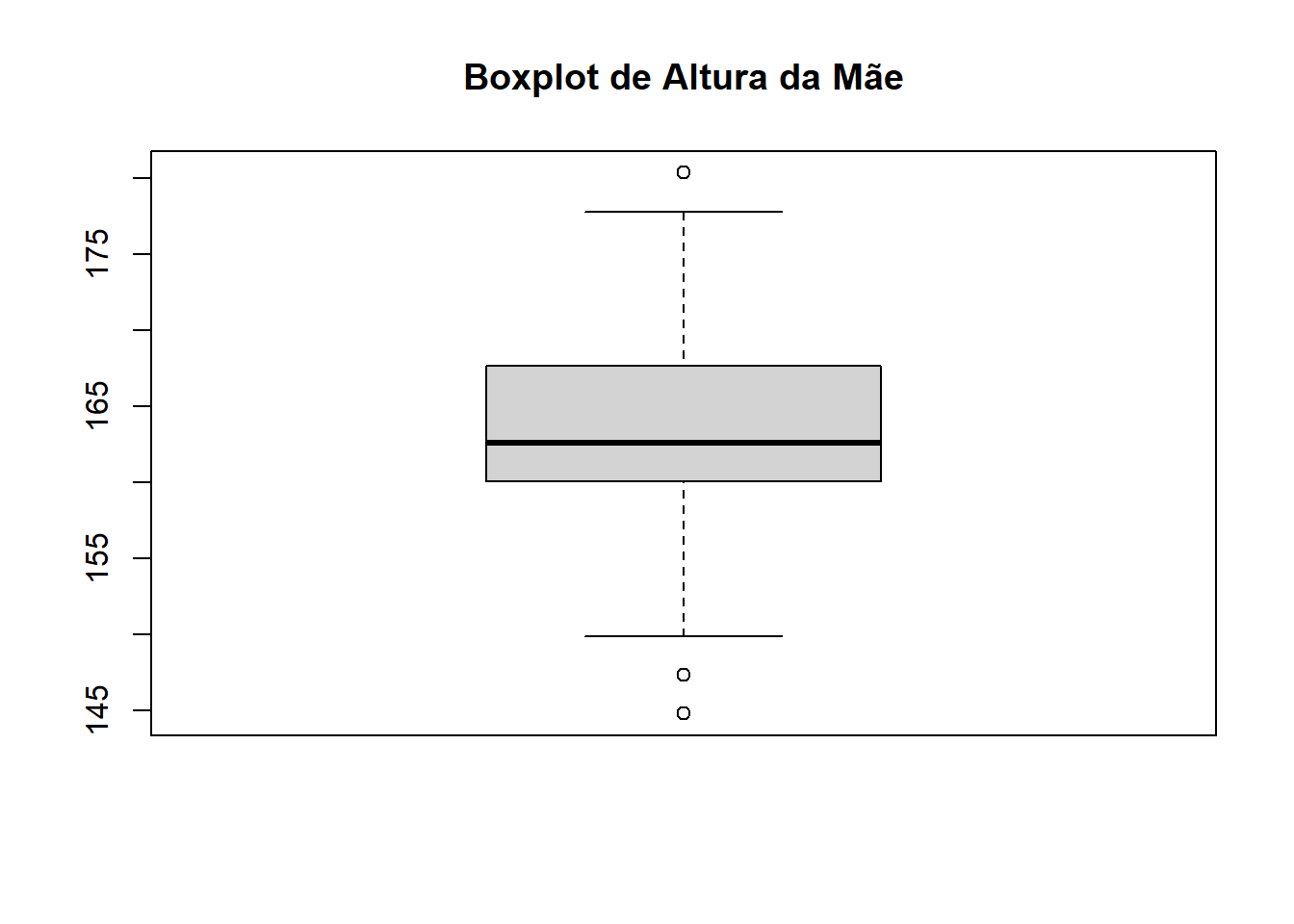
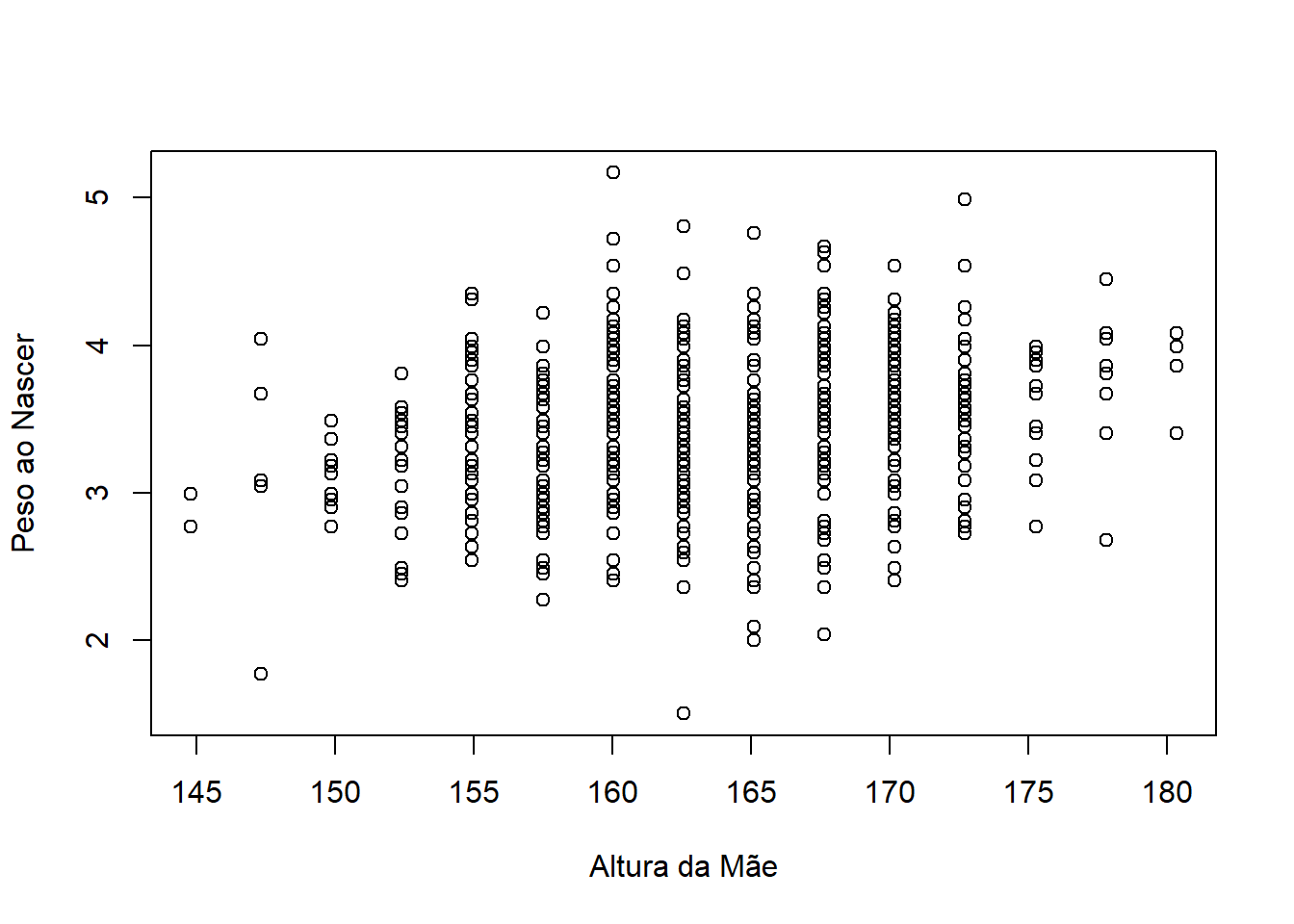
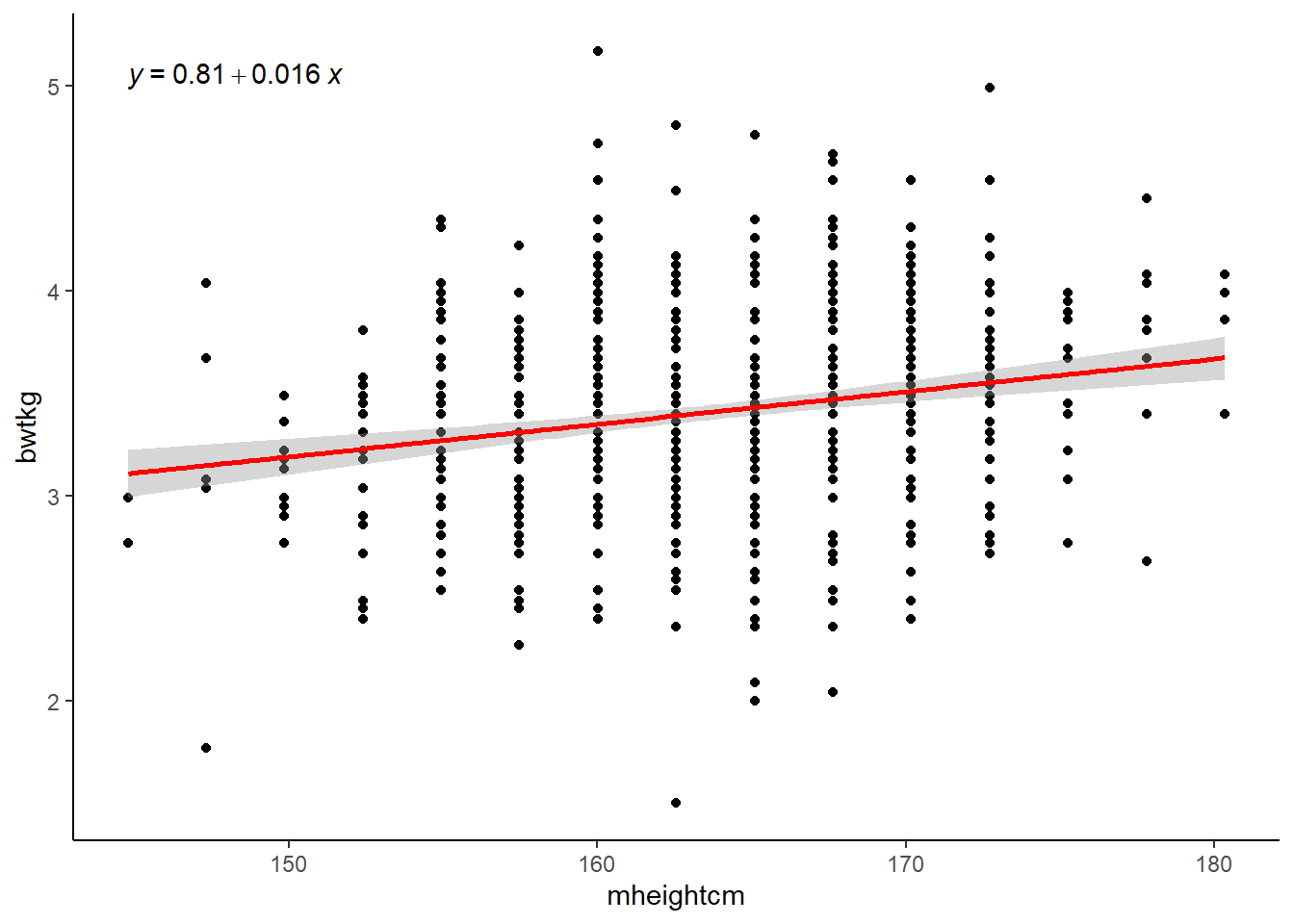
$ mheightcm <dbl> 167.64, 160.02, 154.94, 172.72, 170.18, 160.02, 165.10, 162…
$ mweightkg <dbl> 63.50, 58.97, 57.15, 68.04, 50.80, 59.42, 60.78, 56.70, 64.…
$ mcig <dbl> 25, 0, 0, 2, 17, 0, 0, 17, 0, 0, 25, 0, 25, 17, 0, 0, 0, 0,…
$ fageyrs <dbl> 37, 35, 38, 30, 28, 34, 26, 29, 32, 41, 26, 27, 46, 38, 30,…
$ fheightcm <dbl> 187.96, 180.34, 165.10, 185.42, 180.34, 167.64, 180.34, 180…
$ fedyrs <dbl> 12, 10, 12, 16, 10, 12, 12, 12, 14, 16, 16, 12, 16, 16, 16,…
$ fcig <dbl> 25, 7, 17, 7, 17, 17, 0, 7, 0, 0, 25, 2, 0, 17, 0, 2, 12, 0…
$ lowbwt <dbl> 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…